MRD技术系列解析(三):背景噪音干扰
MRD技术系列解析(三):背景噪音干扰

前言
在之前的几期《MRD技术系列解析》科普专题中,我们探讨了单点突变灵敏度和监测突变数量与MRD性能之间的关系,这两方面主要影响了检测的灵敏度。而在实践过程中,临床不仅关心是否能早期发现肿瘤来源的分子异常,同时也会关注检测到的异常信号是否真实来源于肿瘤。因此,除了灵敏度,关于MRD监测的另一关键评估指标——特异性也至关重要。
一般来说,测序过程中引入的随机错误及克隆性造血等背景噪音会严重干扰MRD判读进而降低特异性。那这些背景噪音是如何干扰MRD判读或者说特异性的?我们又该如何规避其影响?本篇文章将为您一一解答。
浅谈炙手可热的MRD技术
MRD技术系列解析(一):单点突变灵敏度
MRD技术系列解析(二):监测突变数量
MRD技术系列解析(三):背景噪音干扰
如何快速识别MRD产品性能
背景噪音包括哪些因素?
1、PCR、测序过程引入的随机错误
在文库构建的PCR过程中,DNA聚合酶会引入一些原始样本基因组上不存在的随机错误;另外,在测序上机前的文库扩增环节中,因扩增偏好性存在也会造成某些区域扩增效率更高,而另一些区域则被低效覆盖,带来后续测序结果的偏差。以常用的illumina测序仪为例,根据不同的测序读长、base calling算法以及检测的突变类型不同,测序错误率约在1%~0.05%[1]。当存在这些系统错误时,通过加大测序深度来提高MRD检出率的方法往往会引入更多随机错误,导致难以区分所检测到的突变是肿瘤样本真实突变还是系统错误所致的假阳性。那么哪些情况下,我们需要特别警惕临床实验中的随机错误呢?简单来说,样本质量差导致的检测背景复杂、高深度检测微量靶标伴随的高背景噪音,都容易受到PCR与测序过程带来的假阳性影响。因此MRD监测在提高测序深度的同时,需格外注意排除实验随机错误的影响。
2、克隆性造血突变的影响
什么是克隆性造血?克隆性造血(Clonal hematopoiesis,CH),也称潜能未定的克隆性造血(Clonal hematopoiesis of indeterminate potential,CHIP),它是一种常见的衰老相关现象。克隆性造血细胞群携带的一些随机基因突变,比如DNMT3A、TET2和ASXL1等基因突变,通常也会出现在实体瘤患者的血液及肿瘤组织中,干扰肿瘤突变图谱分析结果(图1)[2]。
2019年,《Nature Medicine》杂志上刊发了一篇MSKCC和GRAIL研究团队的重要研究成果。研究指出,肿瘤患者血液中53.2%的cfDNA突变来自血液中的白细胞而非肿瘤细胞,造成这种现象的主要原因就是克隆性造血突变[3]。克隆性造血突变中驱动突变相对少见,因此其对靶向基因的检测影响较小,但是对于从血液中捕获微量肿瘤信号的MRD检测来说,克隆性造血突变的影响则非常巨大,必须通过特定手段和算法来消除。
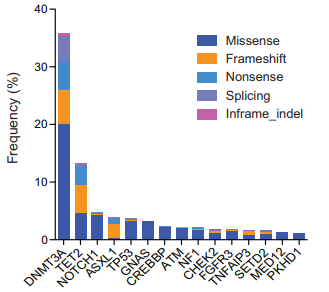
图 1. 主要克隆性造血突变及突变类型[4]
如何规避背景噪音的干扰?
在临床实践中,特异性高就意味着检测到的分子异常大概率真实来源于肿瘤,据此制定的临床决策也更符合患者的疾病状态。而背景噪音的存在会干扰我们对MRD的判读,造成假阳性结果,将患者错误地归为“高危”群体。这不仅增加了患者的心理负担,还可能误导临床进行不必要的强化治疗,造成医疗资源的极大浪费。因此,背景噪音对于MRD判读和治疗决策的制定意义重大。那究竟怎样才能避免背景噪音的干扰呢?
解决方法1:引入分子标签,识别扩增与测序过程中的随机错误
通常情况下,检测公司会通过UMI(分子标签)来追踪真实的ctDNA突变,以降低测序流程中随机错误的干扰。如图2所示,通过分子标签的标记,样本中的每一个初始DNA片段都结合了特异的分子标签序列,这些序列会随目标序列一起经过文库构建、PCR扩增并被一同测序。在最终测得的序列中,凡带有相同分子标签的序列,即可说明它们来源于同一条原始DNA片段;若带有不同分子标签,则提示它们来自不同的原始DNA片段。如果测得的突变仅出现于同一原始DNA的部分序列,则说明该突变是在PCR和测序过程中随机发生的,并非真突变。据此,我们就可以在去除冗余的过程中,将PCR和测序等过程带来的系统突变识别出来并进行剔除[5]。
世和在2016年推出了自主研发的ATG-seq三重降噪技术(Automated Triple Groom Sequencing)。该技术结合三种独立开发的优化系统(BBAM、DDAM、BISC),分别通过单分子标签去除PCR随机错误、双分子标签还原DNA双链去除单链损伤错误;再通过生信算法抛光降低背景噪音,实现cfDNA测序背景的大幅度纯化。

图 2. 世和基因ATG-seq技术去除PCR及测序随机错误
解决方法2:通过白细胞对照、健康人数据库比对等多重机制过滤克隆性造血突变
世和基因于2015年率先在国内推出肿瘤液体活检产品,在产品研发阶段就意识到CH对肿瘤特有突变判读的潜在影响。2020年,世和基因联合国内顶尖临床、分子专家团队,对4500多例不同肿瘤患者的CH突变进行了系统研究,并揭示了CH突变的分子特征[4]。研究发现,CH突变丰度较低,在健康人和肿瘤患者之间通常没有显著差异;但CH突变数量与年龄的增长呈正相关(如图3),且CH突变具有个体特异性,可以通过配对白细胞样本进行对比进而过滤。
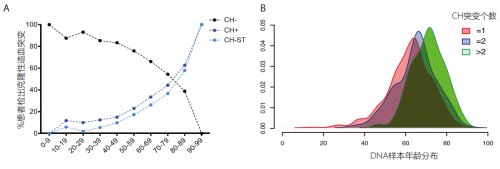
图 3. 克隆性造血与年龄呈正相关[4]
世和基因在MRD临床检测中,首先会进行严格的白细胞对照检测和中国人群CH数据库比对;其次,我们还会对患者基线组织进行突变配对分析;最后再结合患者年龄和多次动态监测综合评估,力求尽可能排除CH干扰,精准识别肿瘤特有突变。
结语
总的来说,在提高MRD检测特异性上,世和基因基于ATG-seq液体活检技术,结合白细胞对照、组织样本配对以及健康人克隆性造血突变数据库等过滤机制进行MRD综合判定,从而有效规避背景噪音的干扰。
MRD产品的灵敏度、特异性等性能受到大量因素的影响,而市面上MRD检测产品五花八门,性能良莠不齐,应该如何快速识别正确选择呢?我们将在《MRD技术系列解析》科普专题的下一篇中,围绕这一话题进行深入探讨,敬请期待。
参考文献
[1] Kinde et al. Detection and quantification of rare mutations with massively parallel sequencing. Proc Natl Acad Sci U S A. 2011 Jun 7;108(23):9530-5.
[2] Steensma et al. Clonal hematopoiesis of indeterminate potential and its distinction from myelodysplastic syndromes. Blood. 2015 Jul 2;126(1):9-16.
[3] Razavi et al. High-intensity sequencing reveals the sources of plasma circulating cell-free DNA variants. Nat Med. 2019 Dec;25(12):1928-1937.
[4] Li et al. Comprehensive next-generation profiling of clonal hematopoiesis in cancer patients using paired tumor-blood sequencing for guiding personalized therapies. Clin Transl Med. 2020 Nov;10(7):e222.
[5] Schmitt et al. Detection of ultra-rare mutations by next-generation sequencing. Proc Natl Acad Sci U S A. 2012 Sep 4;109(36):14508-13.
发表评论




暂时没有评论,来抢沙发吧~